Resumen
Resumen de tema de trabajo
Obtenemos, analizamos e integramos datos x-ómicos de patógenos de interés en salud humana y animal mediante herramientas bioinformáticas y de biología molecular.
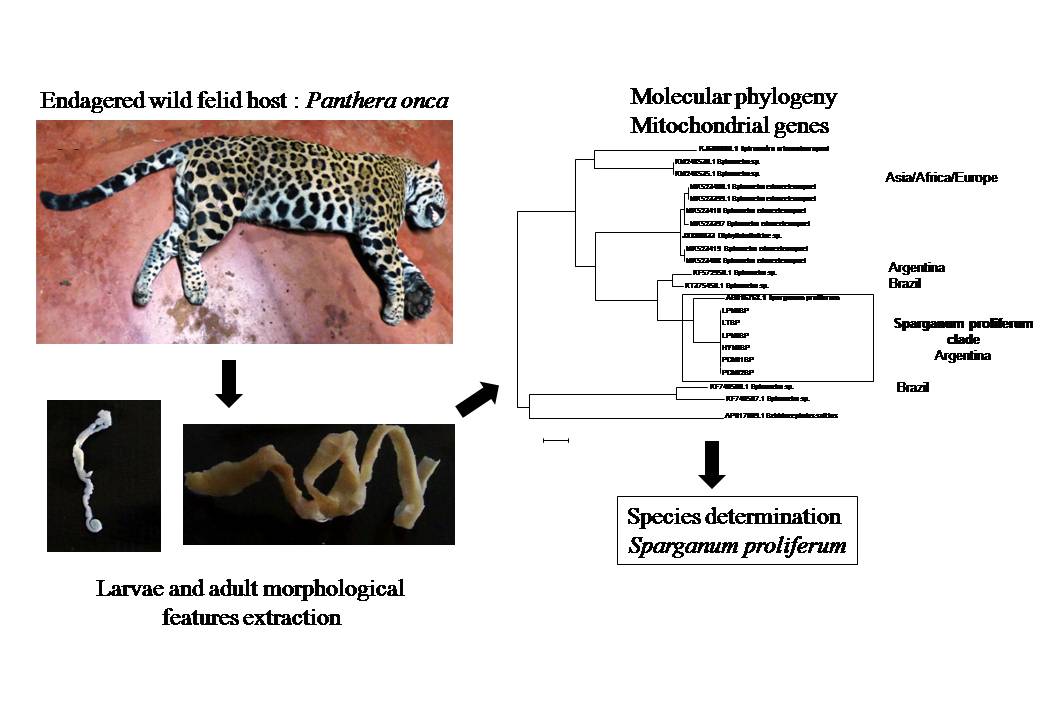
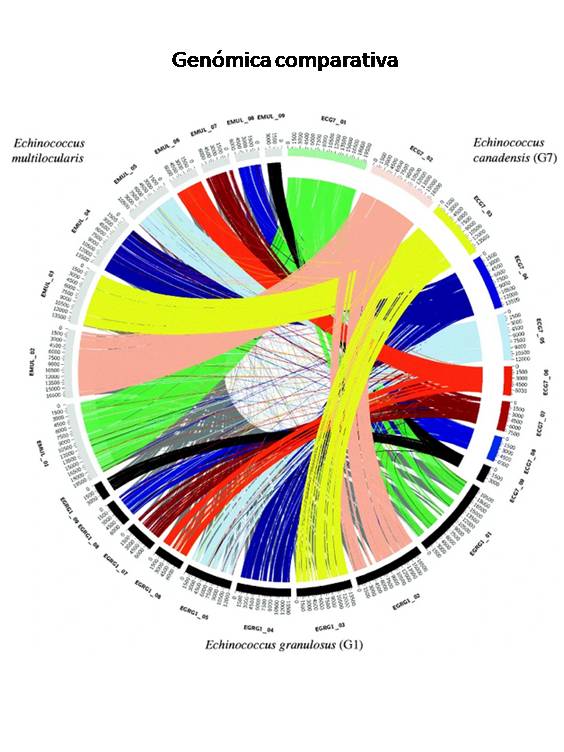
Una de las principales líneas de trabajo se enfoca en los helmintos parásitos (nematodos, cestodos, trematodos). Estos organismos provocan enfermedades graves y difíciles de tratar en humanos, animales y plantas. Un alto porcentaje de la población de la Tierra, principalmente en los países en desarrollo, sufre de infecciones por helmintos. En América Latina, las estimaciones de prevalencia son ~ 30%. El campo de la helmintología se vio profundamente beneficiado por las tecnologías de secuenciación de alto rendimiento que permitieron la confirmación de todo el genoma y la expansión del conocimiento previo sobre genes y vías particulares importantes para la supervivencia de los parásitos, como los reportados para cestodos, proyectos en los que hemos participado (PMID: 23485966) o nos encontramos ejecutando (PMID: 28241794, PMID: 31440275). Con la generación de secuencias completas del genoma descubrimos que una alta proporción del genoma de parásitos están compuestos de genes sin homología en especies modelo (PMID: 29249363), lo que representa un gran interrogante en términos de su relevancia para el desarrollo y supervivencia de estos parásitos. El siguiente paso en esta caracterización a nivel genómico implica el estudio de la interacción de productos génicos de parásitos mediante estrategias de genómica funcional. En el caso de los parásitos, la genómica funcional tiene dos facetas, ya que sus productos génicos pueden interactuar tanto con las moléculas del parásito como con las moléculas del hospedador, en ese sentido hemos estudiado varias proteínas y ARNs pequeños no codificantes específicos del parásito que interactuarían con el hospedador.
Además trabajamos transversalmente y demanera interdisciplinaria con los grupos de investigación del iB3 y diversos institutos dentro y fuera del país.
Líneas de investigación
Utilizamos herramientas bioinformáticas y bases de datos especializadas para el estudio de enfermedades poco frecuentes en humanos, utilizando como modelos a la rata (en colaboración con https://ib3.fbmc.fcen.uba.ar/project/genomica-y-diseno-de-sistemas-biologicos-ld/) y el protista ameboide Dictyostelium discoideum (en colaboración con https://ib3.fbmc.fcen.uba.ar/project/desarrollo-y-morfogenesis-en-dictyostelium-discoideum/). En estos proyectos la información proveniente de genomas completos es utlizada para determinar el grado de conservación de los genes involucrados en las distintas EPOF con los modelos de laboratorio mencionados así como para la indetificación de secuencias determinantes de la regulación génica . La integración de los análisis de secuencias de ADN y expresión con la información médica existente nos permite indetificar con mayor precisión los determinantes genéticos de estas enfermedades. Además para la generación de nuevos datos x-ómicos nos encontramos incorporando recursos de secuenciación de tercera generación (Oxford Nanopore).
La llamada secuenciación paralela masiva o de siguiente generación (next generation sequencing, NGS), estableció un salto de varios órdenes de magnitud en la cantidad de genes que pueden ser estudiados en paralelo. Mediante esta metodología se puede interrogar el genoma completo de un organismo, la secuencia codificante del mismo o exoma, o bien un conjunto de genes de interés (panel de genes o secuenciación paralela masiva dirigida). También es posible analizar todos los genes que se están expresando en un tipo celular o condición, lo que se conoce como transcriptómica, o realizar estudios del conjunto de microorganismos en muestras biológicas (metagenómica) y su expresión (metatranscriptómica) sin necesidad de aislarlos o cultivarlos. En la actualidad existen varias plataformas y desarrollos tecnológicos para estos estudios masivos. La Plataforma de Secuenciacion Oxford Nanopore de iB3 se basa en nanoporos sensibles al paso del ADN/ARN. Esta tecnología permite la secuenciación de la molécula de ADN o ARN de manera “nativa”, sin necesidad de amplificar previamente ni generar intermediarios de secuenciación con la posibilidad de obtener secuencias largas (de 10.000 a 50.000 nucleótidos). La generación de secuencias de mayor longitud permite ensamblar los genomas, metagenomas y transcriptomas de una manera eficiente, acortando los tiempos computacionales y simplificando los algoritmos de ensamblaje. Además permite resolver regiones de las secuencias que poseen motivos “repetidos”, es decir, cuya composición de nucleótidos tiene baja complejidad y no es posible de ensamblar con las metodologías de secuenciación que generan fragmentos cortos. Asimismo permite la secuenciación en tiempo real y la determinación de metilaciones. Nuestra plataforma permite realizar estudios genómicos, metagenómicos, transcriptómicos y metatranscriptómicos mediante secuenciación paralela masiva de tercera generación. Nuestras líneas de investigación aplican esta tecnología a estudios de salud humana, animal y ambiental, utilizando el enfoque Una Sola Salud. Se cuenta con equipamiento y profesionales de múltiples disciplinas que generan sinergia entre grupos de trabajo y potencian el desarrollo de herramientas biotecnológicas.
Integrantes:
Natalia Macchiaroli
macchiaroli.natalia@gmail.com
Investigadora Asistente CONICET
Lucas Federico Arce
lucasfarce.lfa@gmail.com
Becario doctoral ANPCyT
Ines Sananez
ines.sananez@gmail.com
Becaria posdoctoral CONICET
Juan Pablo Arrabal
jparrabal.vet@gmail.com
Becario doctoral UNL-INMet
Marina Luz Ingravidi
maru.luz98@hotmail.com
Becaria Doctoral CONICET
Kevin Calupiña
davidcalupina08@gmail.com
Estudiante de Maestria en Biología Molecular UBA
Notas de divulgación
- https://www.conicet.gov.ar/mapa-genetico-de-la-tenia-una-ruta-hacia-la-cura/.
- https://www.agenciacyta.org.ar/2016/06/avance-contra-una-grave-enfermedad-que-transmiten-los-perros/.
- https://www.youtube.com/watch?v=QCCIIjwx_fY
- “https://m24digital.com/cientificos-argentinos-avanzan-en-el-tratamiento-de-la-hidatidosis-una-enfermedad-grave-transmitida-por-perros/.
Publicaciones Seleccionadas
- https://www.ncbi.nlm.nih.gov/pubmed/?term=Kamenetzky+l
Colaboradores
NACIONALES
Centro de Investigación en Señales, Sistemas e Inteligencia sinc(i), UNL
>Dr. Diego Milone
>Dra. Georgina Stegmayer
ANLIS-Dr. Carlos G. Malbrán
>Dra. Silvana Carnevale
Instituto de Investigaciones Bioquímicas de La Plata (INIBIOLP), UNLP
>Dra. Gisela Franchini
FVET, UNL
>Dr. Pablo Beldoménico
INBIRS-FMED-UBA
>Dra. Gabriela Turk
>Dr.Daniel Salomón
>Dr. Jorge Geffner
Nanobiotec-FFyB-UBA
> Dr. Alejandro Nusblat
IMPAM-FMED-UBA
>Dra. Marcela Cucher
>Dra. Mara Rosenzvit
>Dra. Maria Luján Cuestas
INTERNACIONALES
Wellcome Sanger Institute, UK
>Dr. Mark Blaxter
ITV, Vale, Brasil
>Dr. Guilherme Oliveira
Tartu University, Estonia
>Dr. Urmas Saarma
Universidad de la República, Uruguay
>Dr. Pablo Smircich
Instituciones/Agencias Financiadoras:
UBA, CONICET, ANPCyT, Ministerio de Educación, Perez-Guerrero, Royal Society.