Resumen
En el laboratorio utilizamos diferentes levaduras 1) como modelo experimental para estudiar las bases moleculares de las enfermedades congénitas de glicosilación humanas, focalizando en los mecanismos de N-glicosilación de proteínas involucrados y 2) como plataforma para procesos biotecnológicos: producción de proteínas recombinantes y otros metabolitos de interés en la industria farmacológica, en biorremediación y en bioenergética.
Líneas de investigación
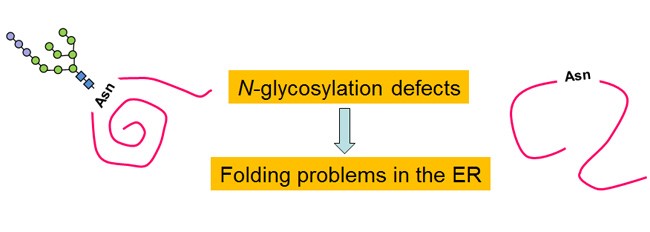
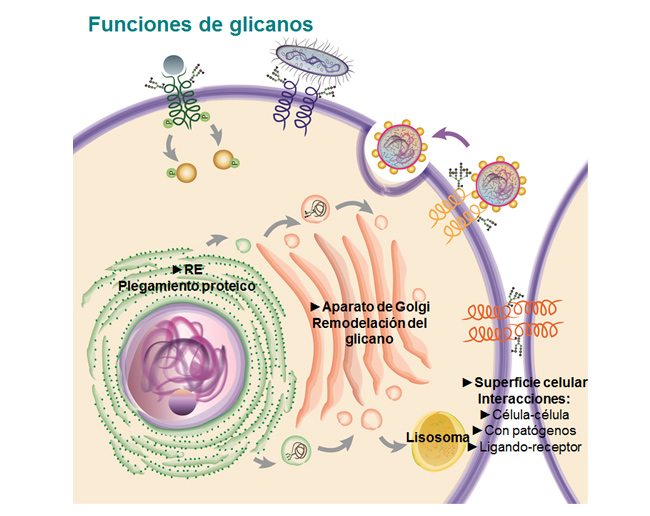
Se conocen actualmente más de 80 enfermedades hereditarias humanas debidas a defectos en la N-glicosilación de proteínas. Las CDG Tipo I se deben a defectos durante la transferencia de glicanos a las proteínas y las CDG tipo II son debidas a defectos en la remodelación posterior de los glicanos. Los pacientes presentan fallas multisistémicas que aparecen a diversas edades. Las CDG IIb o MOGS-CDG son causadas por mutaciones en la glucosidasa I (GI), una proteína de membrana del RE responsable del primer paso del procesamiento de los N-glicanos. En el laboratorio estudiamos las bases moleculares de la enfermedad usando levaduras S. pombe mutantes que simulan estos defectos y exploramos las causas celulares de los defectos producidos por la falta de GI, la búsqueda de funciones adicionales de esta enzima y evaluamos posibles tratamientos de CDG tipo IIb.
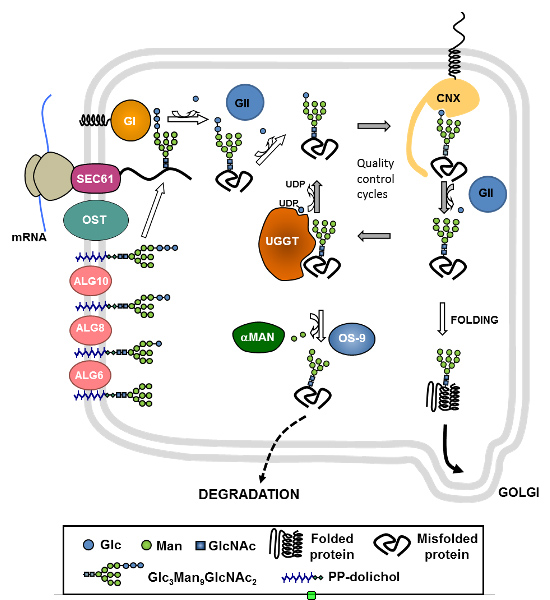
Las levaduras son ampliamente utilizadas en la industria alimenticia, como modelo de estudio de células eucariontes y como plataforma de expresión de proteínas recombinantes. Son hongos unicelulares, muchos de ellos no patógenos y con gran cantidad de herramientas disponibles para hacer estudios bioquímicos y manipulaciones genéticas. En el laboratorio diseñamos levaduras Saccharomyces cerevisiae como plataforma para procesos de biorremediación de desechos de la industria láctea acoplados a la producción de bioetanol, y también como organismo para producción heteróloga de un compuesto fotoprotector. También utilizamos la levadura Pichia pastoris como plataforma para la producción recombinante de proteínas de interés biotecnológico, como el dominio RBD de Sars-CoV2 para su uso en kits diagnóstico o candidato vacunal, o nanoanticuerpos para ser usados en reemplazo de anticuerpos secundarios entre otras.
Integrantes:
Sofía Orioli
sorioli.97@gmail.com
Lic. en Biotecnología
Becaria Doctoral CONICET
Victoria Albeck
victoriaalbeck@gmail.com
Becaria UBA
Tesinista de Lic. en Ciencias Biológicas
Fiorella Iannello Vitale
fioreiannello@hotmail.com
Pasante/Tesinista Ingenieria Industrial
LinkedIn
Notas de divulgación:
Oficina de Vinculación Tecnológica de la FCEN, UBA. Transmitido por YouTube (2021)
Exactas y el Covid-19: Innovación y transferencia
Programa televisivo “Noche de Mente”, TV Publica, 2023
Biotecnología de levaduras
Radio Fénix, FM 100.3, Ciudad de Buenos Aires
Desarrollo de vacuna Argenvac
NexCiencia – Noticias de Ciencia y Técnica
Avance para frenar la pandemia- Proteína buena, barata y abundante
Colaboradores:
Dr. Guillermo Alonso
INGEBI
Dr. Nancy Dahms
Medical College Wisconsin
Dr. Diego Libkind
Universidad del Comahue
Dr. Itatí Ibañez
INQUIMAE
Publicaciones Representativas:
- Idrovo-Hidalgo T, Pignataro MF, Bredeston L, Elias F, Herrera MG, Pavan MF, Foscaldi S, Suireszcz M, Fernández NB, Wetzler DE, Paván CH, Craig PO, Roman EA, Ruberto LAM, Noseda DG, Ibañez LI, Czibener, C, Ugalde, JE, Argentinian AntiCovid Consortium, Nadra AD, Santos J, D’Alessio C (2024)
Deglycosylated RBD produced in Pichia pastoris as a low-cost sera COVID-19 diagnosis tool and a vaccine candidate
Glycobiology 34, cwad089
DOI: 10.1093/glycob/cwad089 - Gallo G, Valko A, Herrera Aguilar, N, Weisz, AD y D’Alessio, C (2022)
A novel fission yeast platform to model N-glycosylation and the bases of congenital disorder of glycosylation Type I
Journal of Cell Science
DOI: 10.21769/BioProtoc.4508 - Argentinian AntiCovid Consortium (2022): Berguer PM, Blaustein M, Bredeston LM, Craig PO, D’Alessio C, Elias F, Farré P, Fernández NB, Gentili HG, Gándola YB, Gasulla J, Gudesblat, GE, Herrera MG, Ibañez LI, Idrovo-Hidalgo T, Nadra AD, Noseda DG, Paván CH, Pavan MF, Pignataro MF, Roman EA, Ruberto LAM, Rubinstein N, Sanchez MV, Santos J, Wetzler DE, Zelada AM
Covalent coupling of Spike’s receptor binding domain to a multimeric carrier produces a high immune response against SARS-CoV-2
Scientific Reports 12, 692
DOI: 10.1038/s41598-021-03675-0 - Argentinian AntiCovid Consortium (2020): Arbeitman CR, Auge G, Blaustein M, Bredeston LM, Corapi ES, Craig PO, Cossio LA, Dain L, D’Alessio C, Elias F, Fernandez NB, Gasulla J, Gorojovsky N, Gudesblat GE, Herrera MG, Ibañez LI, Idrovo-Hidalgo T, Iglesias Rando M, Kamenetzky L, Nadra AD, Noseda DG, Pavan CH, Pavan MF, Pignataro MF, Roman EA, Ruberto LAM, Rubinstein N, Santos J, Velazquez F, and Zelada AM
Structural and Functional Comparison of SARS-CoV-2-Spike Receptor Binding Domain Produced in Pichia pastoris and Mammalian Cells
Scientific Reports 10, 21779
DOI: 10.1038/s41598-020-78711-6 - Gallo, GL; Valko, A; Aramburu, SI; Etchegaray, E; Völker, C; Parodi, AJ y D’Alessio, C (2018)
Abrogation of glucosidase I-mediated glycoprotein deglucosylation results in a sick phenotype in fission yeasts: Model for the human MOGS-CDG disorder
Journal of Biological Chemistry (293) 19957- 19973
DOI: 10.1074/jbc.RA118.004844 - Bredeston, LM; Marino-Buslje, C; Mattera, VS; Buzzi, LI; Parodi AJ y D’Alessio C (2017)
The conundrum of UDP-Glc entrance into the yeast ER lumen
Glycobiology 27, 64-79
DOI: 10.1093/glycob/cww092 - D’Alessio, C y Dahms, NM (2015)
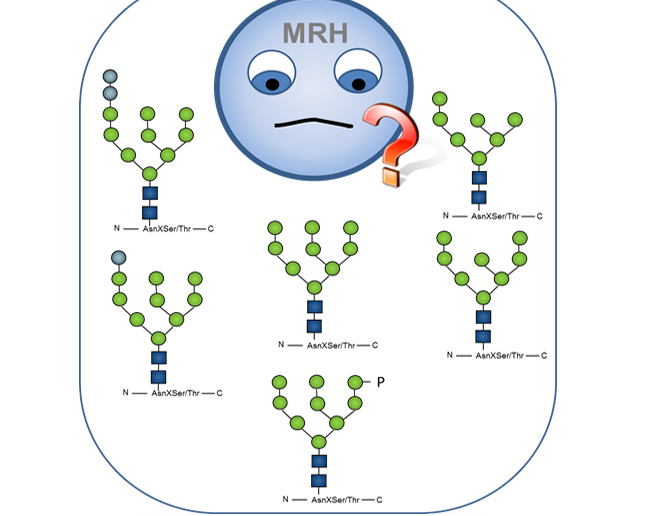
Glucosidase II and MRH-domain containing proteins in the secretory pathway
Current Protein & Peptide Science. 16(1):31-48
DOI: 10.2174/1389203716666150213160438 - Olson, LJ; Orsi, R; Alculumbre, SG; Peterson, FC; Stigliano, ID; Parodi, AJ; D’Alessio, C y Dahms, NM (2013)
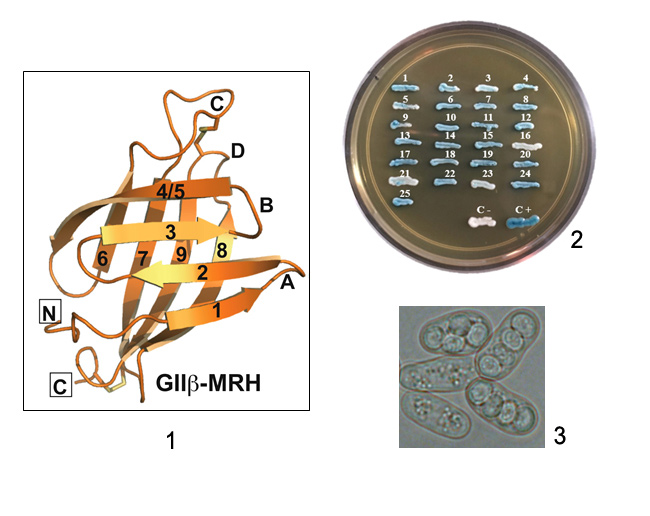
Structure of the Lectin MRH Domain of Glucosidase II, an Enzyme that Regulates Glycoprotein Folding Quality Control in the Endoplasmic Reticulum
Journal of Biological Chemistry 288(23):16460-75
DOI: 10.1074/jbc.M113.450239 - Stigliano, ID; Alculumbre, SG; Labriola, CA; Parodi, AJ y D’Alessio, C (2011)
Glucosidase II and N-glycan mannose content regulate the half-lives of monoglucosylated species in vivo
Molecular Biology of the Cell 22, 1810-1823
DOI: 10.1091/mbc.e11-01-0019 - C D’Alessio, JJ Caramelo, AJ Parodi (2010)
UDP-Glc:glycoprotein glucosyltransferase-glucosidase II, the ying-yang of the ER quality control
Semin Cell Dev Biol. 21, 491-9
DOI: 10.1016/j.semcdb.2009.12.014 - Stigliano, ID; Caramelo, JJ; Labriola, CA; Parodi, AJ y D’Alessio, C (2009)
Glucosidase II beta subunit modulates N-glycan trimming in fission yeast and mammals
Molecular Biology of the Cell 20(17):3974-84
DOI: 10.1091/mbc.e09-04-0316 - Olson, LJ; Orsi, R; Alculumbre, SG; Peterson, FC; Stigliano, ID; Parodi, AJ; D’Alessio, C y Dahms, NM (2013)
Structure of the Lectin MRH Domain of Glucosidase II, an Enzyme that Regulates Glycoprotein Folding Quality Control in the Endoplasmic Reticulum
Journal of Biological Chemistry 288(23):16460-75
DOI: 10.1074/jbc.M113.450239
Las publicaciones con sus links se encuentran en:
https://scholar.google.com.ar/citations?user=E-p3olYAAAAJ&hl=es